



随着植物基因组编辑技术的快速发展,特别是CRISPR-Cas系统的应用,精准植物遗传改良已成为可能。尽管CRISPR-Cas系统在植物基因组编辑中显示出巨大的潜力,但其应用仍面临一些挑战,特别是不同向导RNA(gRNA)的编辑效率差异,增加了编辑事件筛选的难度。传统策略通常依赖于标记基因(如抗生素或除草剂抗性标记)来筛选转基因事件。
然而,这些标记基因并不直接反映目标基因的编辑状态,研究人员需要进一步筛选转化事件,以确定哪些事件实际发生了所需编辑。以往的研究中通过使用代理报告系统来提升编辑效率,但由于分别使用不同的sgRNA来编辑代理报告基因和内源基因,这些系统只能筛选CRISPR-Cas高表达的事件,却未能有效显示内源基因位点的编辑活性,因此迫切需要开发更为集成和有效的方法来筛选编辑事件。
近日,学校生命科学学院张勇教授团队联合园艺园林学院宋洪元教授团队、马里兰大学Yiping Qi教授在Plant Communications上发表了题为Boosting genome editing in plants with single transcript unit surrogate reporter systems的研究论文。该研究工作基于前期构建的单一转录单元CRISPR-Cas(STU-CRISPR)基因组编辑系统(Tang et al., 2016; Tang et al., 2019),构建了基于代理报告系统的STU-SR植物基因组编辑系统,实现了对Cas9敲除编辑、C to T及A to G碱基编辑事件的高效富集,有效拓展了植物基因组编辑工具箱,为精准植物遗传改良提供了强有力的技术支持。
研究者在STU-CRISPR系统基础上,结合包含内源基因编辑位点的筛选标记报告基因,构建了STU-SR-SSA系统。通过使用相同的sgRNA同时针对报告基因和内源基因进行编辑,建立了报告基因编辑与目标基因编辑的直接联系。当报告基因被sgRNA切割后,可通过SSA被修复为有功能的筛选标记基因,从而筛选得到的抗性植株中发生编辑的概率大幅提升。
水稻中的实验结果显示,STU-SR-SSA系统在测试的内源基因位点上均达到了100%的编辑效率,与传统系统相比,编辑效率显著提升。此外,双等位基因编辑效率也得到了提高,OsPDS-sgRNA02和OsDEP1-sgRNA01两个位点的双等位编辑效率均达到100%(图1)。
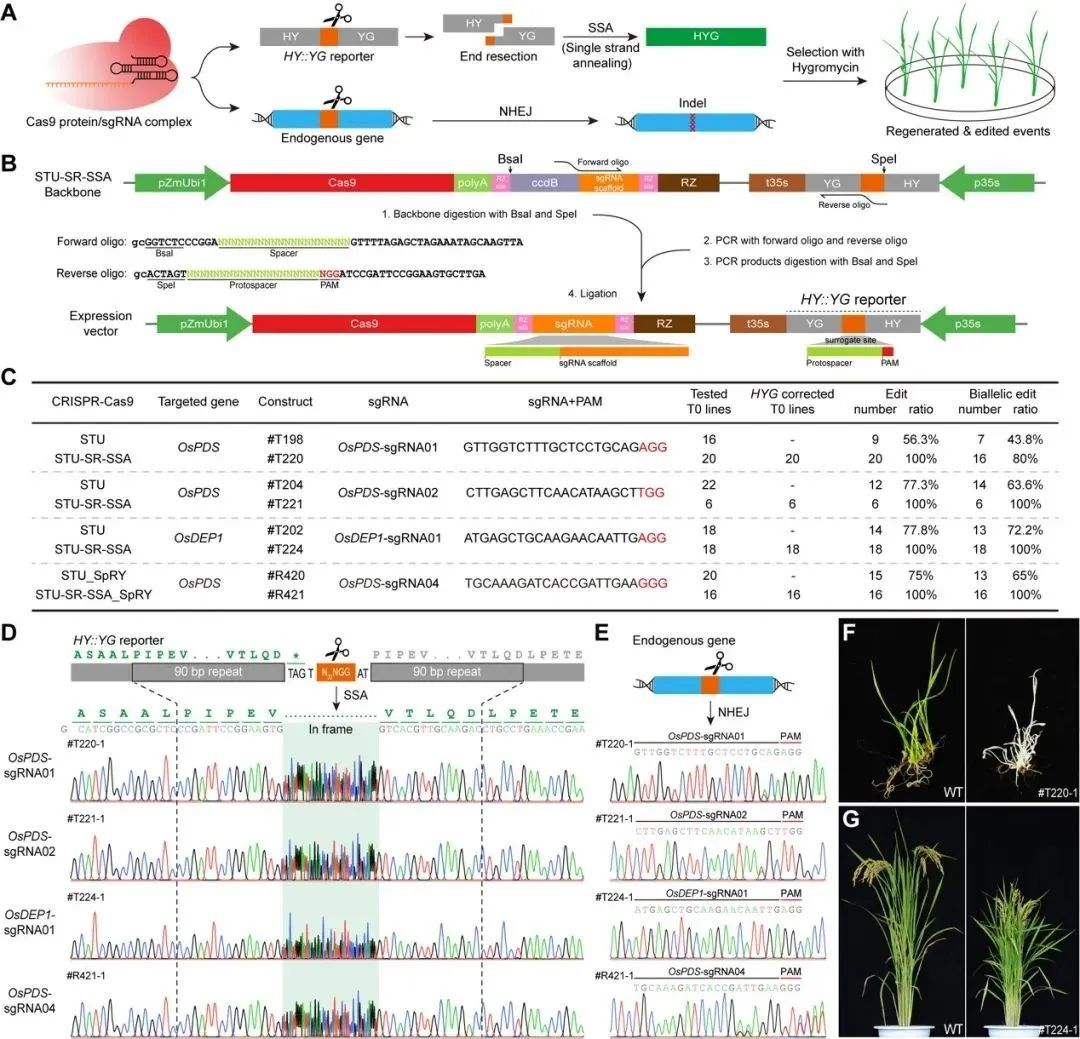
图1 STU-SR-SSA基因组编辑系统构建及编辑活性评价
为实现碱基编辑事件的有效富集,研究者构建了STU-SR-BE系统。通过去除标记基因起始密码子并添加内源基因位点序列得到报告基因,报告基因被C to T或A to G编辑后形成新的起始密码子,恢复标记基因的正常功能。水稻中的结果表明:与传统CRISPR-Cas9系统相比,STU-SR-BE系统在多个内源基因位点上显著提高了编辑效率,与SpRY结合有效拓展了STU-SR-BE系统的应用范围(图2)。
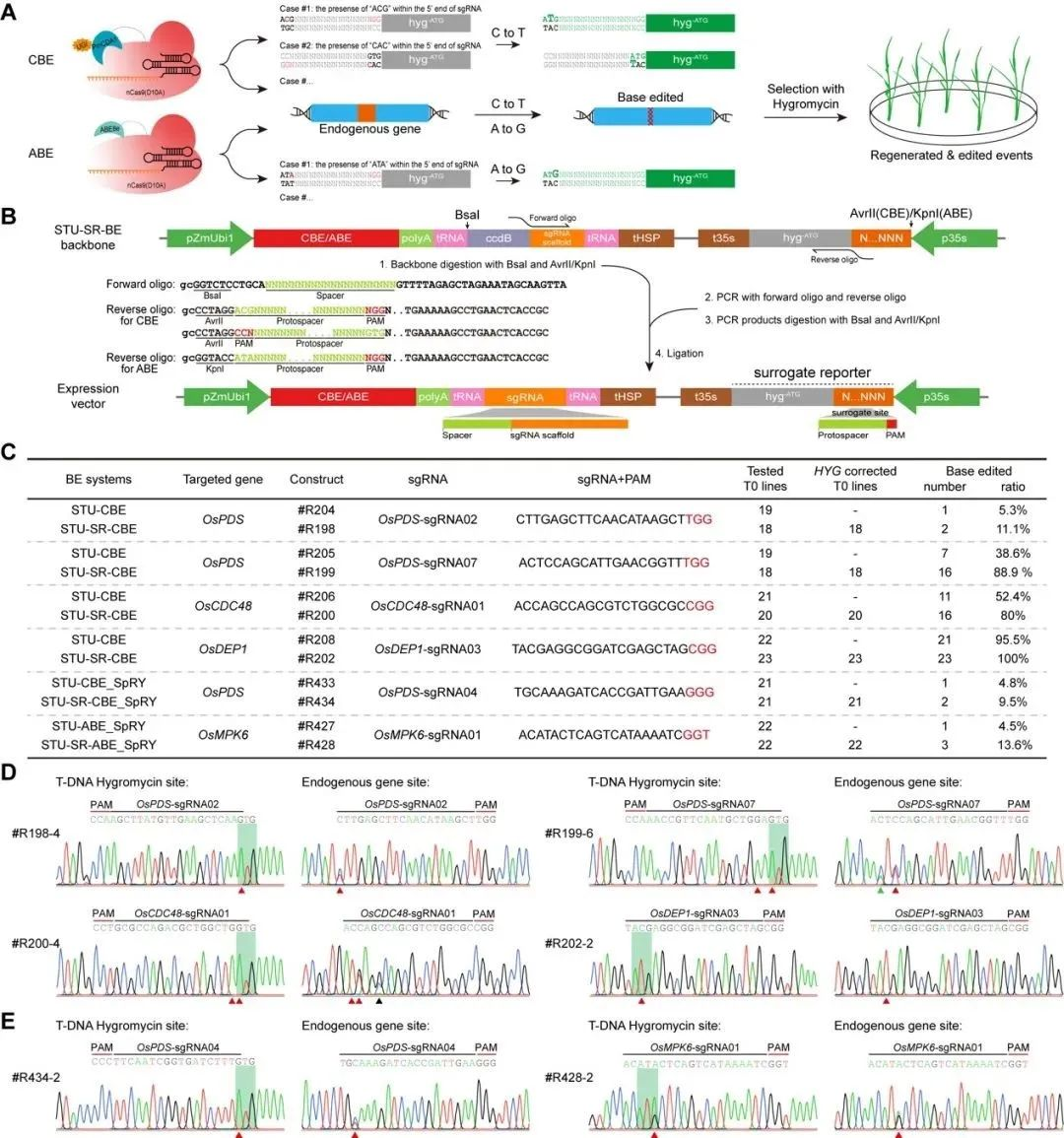
图2 STU-SR-BE基因组编辑系统构建及编辑活性评价
最后,研究者选取双子叶植物甘蓝来测试STU-SR系统的适用性。实验结果表明,与传统的CRISPR-Cas9系统相比,STU-SR-SSA系统在所有测试的内源基因位点上都实现了更高的编辑效率,BoPDS-sgRNA01位点的编辑效率从0%提高到了35%,BoPDS-sgRNA02位点的编辑效率从5%提高到了16.7%,BoBIK-sgRNA01位点的编辑效率显著提高了48.4%(图3)。这表明STU-SR系统能够有效地富集基因编辑事件,并在不同植物物种中具有广泛的应用潜力。
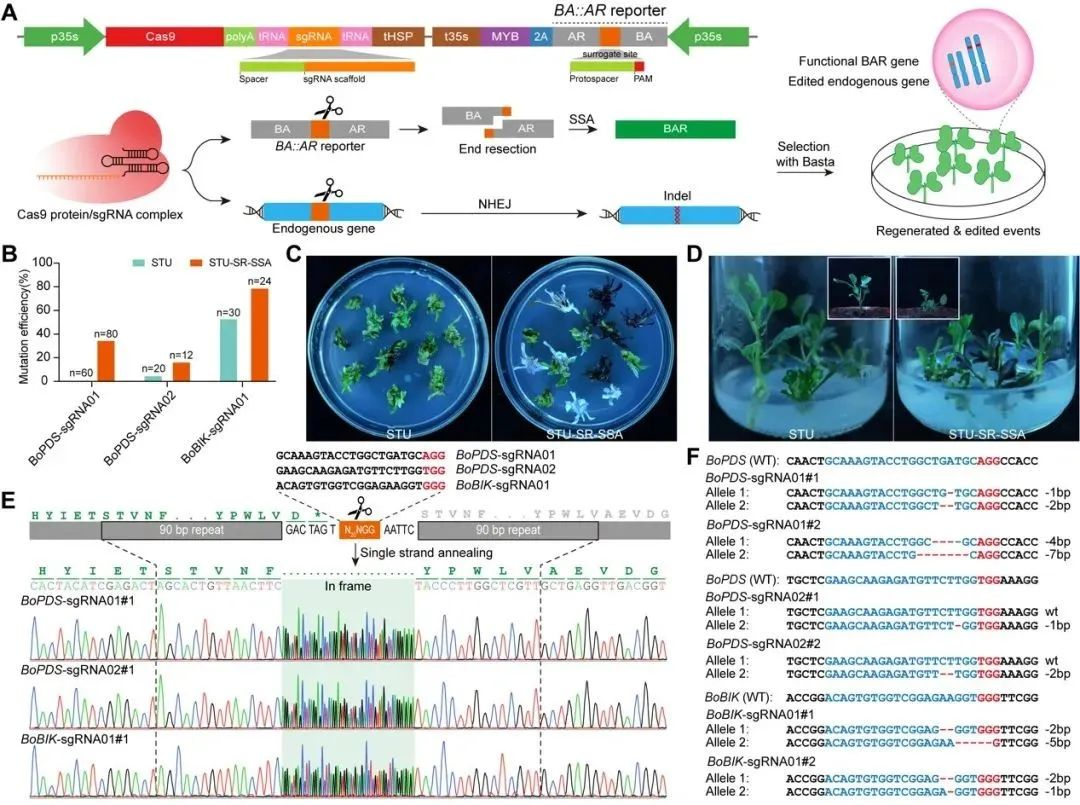
图3 STU-SR-SSA基因组编辑系统在双子叶植物甘蓝中的应用
生命科学学院张勇教授、园艺园林学院宋洪元教授、马里兰大学Yiping Qi教授为论文共同通讯作者,学院唐旭博士、山西大学任秋蓉博士为论文共同第一作者。研究工作得到了国家重点研发计划、国家自然科学基金、四川省科技项目、重庆市技术创新与应用发展项目等项目的资助。
Copyright © 2021 西南大学科学技术处 版权所有
请使用IE10及以上版本或火狐浏览器、谷歌浏览器,以获得最佳浏览效果
渝ICP备05006217号
地址:重庆市北碚区天生路2号西南大学南区行政楼一楼 联系电话/传真:023-68251230 邮编:400715

扫码关注「科技西大」